本記事では、pythonのライブラリである、GEOparseを用いて、GEOのデータを取得・解析する手法について解説します。
GEOとは
NCBIのGEO(Gene Expression Omnibus)は、遺伝子発現情報のデータベースであり、マイクロアレイやRNA-seq実験などで得られたデータが登録されています。
GEOを利用して、実験データや遺伝子発現プロファイルデータを検索・ダウンロードすることができます。
NCBIのウェブサイトでは、データセットの検索やダウンロードが簡単にできます。また、NCBIが提供する「GEO2R」などのツールを利用することで、ブラウザ上でデータをより簡単に解析することができます。
今回は上記のような方法ではなく、pythonのライブラリを用いて、GEOのデータを取得・解析する方法を解説します。
GEOparseのインストール
GEOparseはpipでインストールできます。
pip install GEOparseGEOparseの使い方
GEOparseの公式ドキュメントは以下のサイトから確認できます。
GEOparseでGEOデータセットを取得する
早速、GEOからデータを取得していきます。今回は、GSE221791のデータを使います。
GSEはGene Set Experimentの略で、複数の遺伝子発現データセットをまとめたセットを表します。
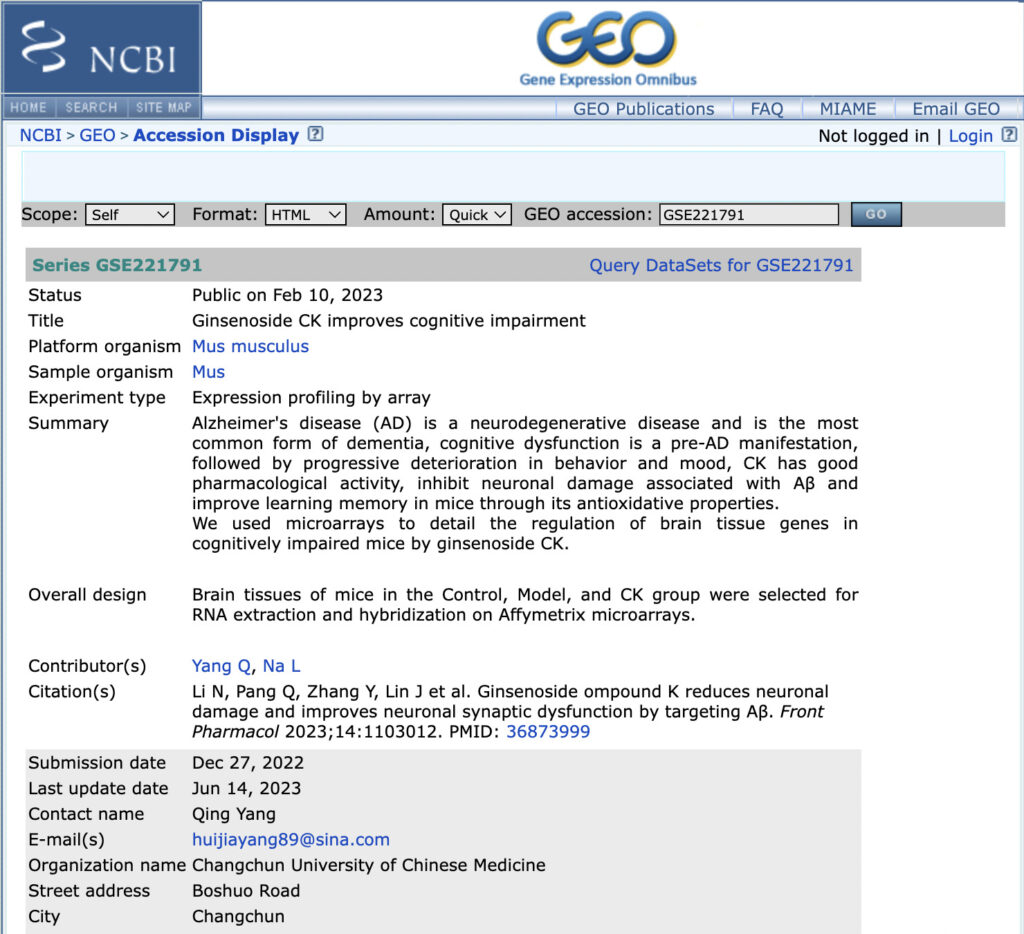
GSE221791は上記の画像のように、アルツハイマー病モデルマウスにCKという薬剤を投与した実験の遺伝子発現をマイクロアレイで取得した結果が登録されています。
GEOparseでデータを取得するには、python上で以下のコマンドを実行します。get_GEOの引数で、geoに取得したいGSE番号、destdirにデータの保存先を指定します。
import GEOparse
gse = GEOparse.get_GEO(geo="GSE221791", destdir="./data")コマンドを実行すると、GSE221791_family.soft.gzがNCBIのサイトからダウンロードされます。
これで下記のSOFT formatted family filesをダウンロードしたことになります。
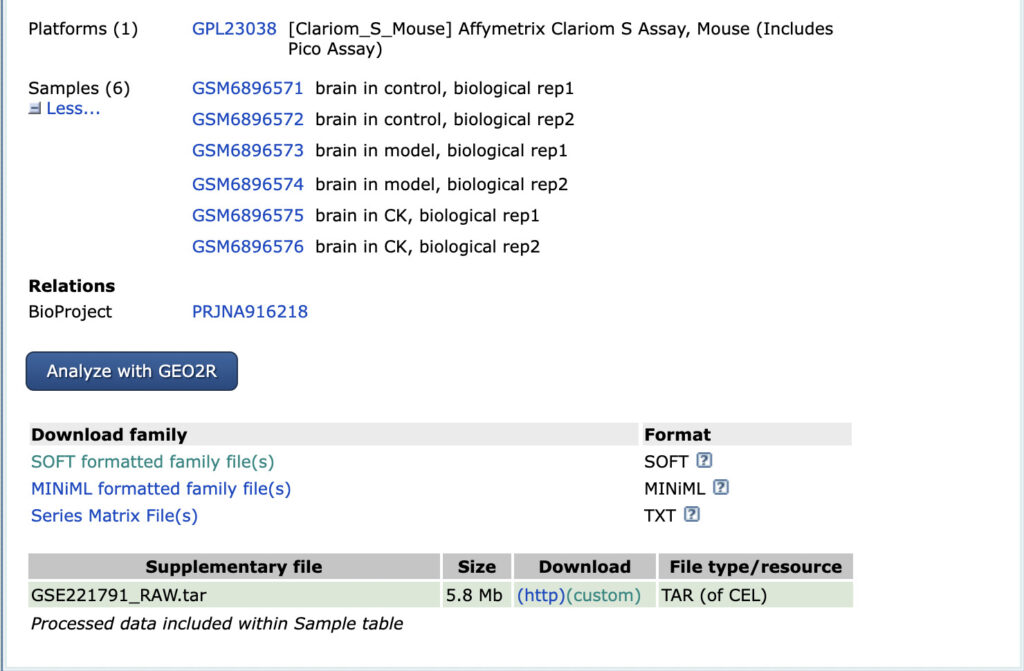
NCBIのサイトから手動でダウンロードしたデータが既にある場合は、destdirにデータの保存場所を指定することでpythonのオブジェクトにパースすることが可能です。
もちろん、手動でNCBIのサイトからダウンロードすることも可能ですが、複数のGSEデータセットをダウンロードしたい場合などは、GEOparseを使うと楽になりそうです。
取得したデータの中身を確認する
pythonのオブジェクトとしてパースされた中身にはたくさんのデータが入っています。
以下のコマンドで中身にどんなデータが入っているかを確認します。
dir(gse)出力を確認すると多くのデータが入っていることを確認できます。
['__class__', '__delattr__',
(中略) ,
'database', 'download_SRA', 'download_supplementary_files',
'geotype', 'get_accession', 'get_metadata_attribute', 'get_type',
'gpls', 'gsms', 'merge_and_average', 'metadata', 'name', 'phenotype_data',
'pivot_and_annotate', 'pivot_samples', 'relations', 'show_metadata', 'to_soft']具体的に見てみましょう。
GPLは、Gene Expression Platformの略で、遺伝子発現データの測定に使用される技術を表します。
GPLには、マイクロアレイやRNA-seqなどのさまざまな技術が登録されています。
print(gse.gpls)出力結果
{'GPL23038': <d: GPL23038>}GSMは、Gene Set Memberの略で、遺伝子発現データセットの中の1つのサンプルを表します。
print(gse.gsms)
print(len(gse.gsms))出力結果
{'GSM6896571': <SAMPLE: GSM6896571>, 'GSM6896572': <SAMPLE: GSM6896572>, 'GSM6896573': <SAMPLE: GSM6896573>, 'GSM6896574': <SAMPLE: GSM6896574>, 'GSM6896575': <SAMPLE: GSM6896575>, 'GSM6896576': <SAMPLE: GSM6896576>}6GSE221791には6つのGSMが含まれていることがわかります。
メタデータをまとめて確認してみます。
from pprint import pprint
pprint(gse.metadata)出力は省略しますが、GSE221791に関する多くのメタデータ情報を確認することができます。
遺伝子発現データを解析する
各サンプルの遺伝子発現量データを表形式でまとめるには以下のコマンドを実行します。
gene_matrix_data = gse.pivot_samples("VALUE")
gene_matrix_data出力結果
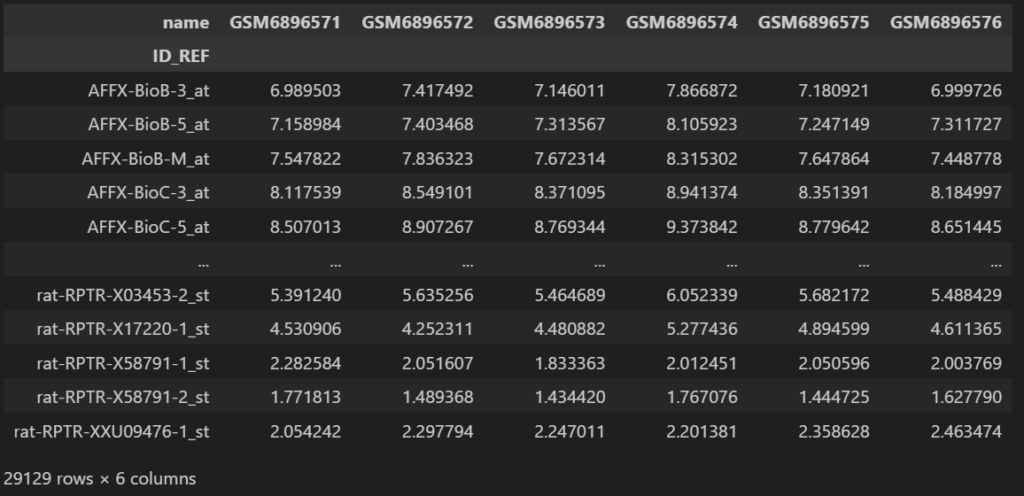
これでプローブIDと各サンプルの遺伝子発現量を示すテーブルデータが得られました。
このテーブルデータを解析することで、複数のサンプル間の遺伝子発現量の違いなどを解析していくことができます。
おわりに
本記事では、pythonのライブラリである、GEOparseを用いて、GEOのデータを取得・解析する手法について解説しました。
ぜひ、pythonを利用した解析も試してみてください。










