バイオインフォマティクスを始めるときに、初心者が最初にぶつかる壁が環境構築です。解析を行うためには非常にたくさんのソフトウェアをインストールして使わないといけないケースが多くあります。
この記事では、簡単にバイオインフォマティクスの解析を始められる環境を準備する方法を解説します。
結論から言うと、自分のPCで手軽にバイオインフォマティクスの解析を始めるには、Dockerを使うのがおすすめです。
windowsの場合は、こちらの記事を参考にしてください。
バイオインフォマティクスを始めるときに、初心者が最初にぶつかる壁が環境構築です。解析を行うためには非常にたくさんのソフトウェアをインストールして使わないといけないケースが多くあります。 この記事では、Windows[…]
Dockerとは
Dockerは「コンテナ」と呼ばれる仮想化技術を作成・実行するためのソフトウェアです。コンテナは隔離されたプロセスなのでホストOSを汚しません。そのため、コンテナを作成したり削除したりすることで、様々なツールを手軽に使うことができます。
Docker Desktopのインストール
Docker Desktopのインストール
WindowsやMacでDockerを使うには、「Docker Desktop」と呼ばれるソフトウェアをインストールします。
Docker Desktopは、インストールが簡単でGUIでも使用できるため、気軽にDockerを使い始めることができます。
本記事では、Mac(Appleシリコン)を前提にインストール方法を紹介します。
macOS Ventura 13.4
Docker Desktop : version 4.19.0
以下のリンクからDocker Desktop for Macをダウンロードします。
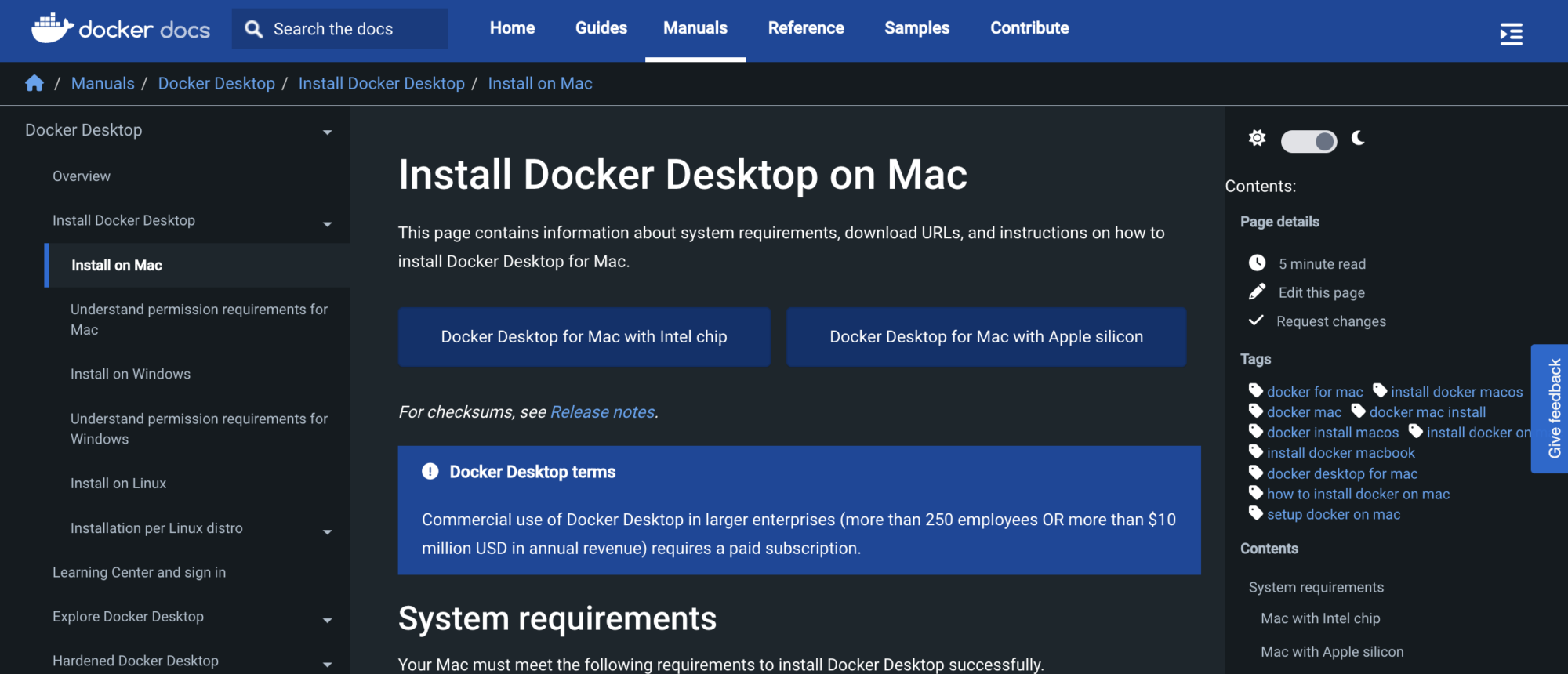
今回はM1チップのMacにインストールするため、「Docker Desktop for Mac with Apple silicon」をクリックし、インストーラーをダウンロードします。
インストーラーをダウンロードできたら以下のような手順で進めてください。
- ダウンロードした「Docker.dmg」をダブルクリック
- Dockerアプリのアイコンをアプリケーションフォルダにドラッグ
- Docker.appを起動(警告が表示されるが「開く」を選択)
- Dockerの規約を確認し、問題なければ「Accept」をクリック
Docker Desktop起動
正常に起動すると以下のような画面が表示されます。
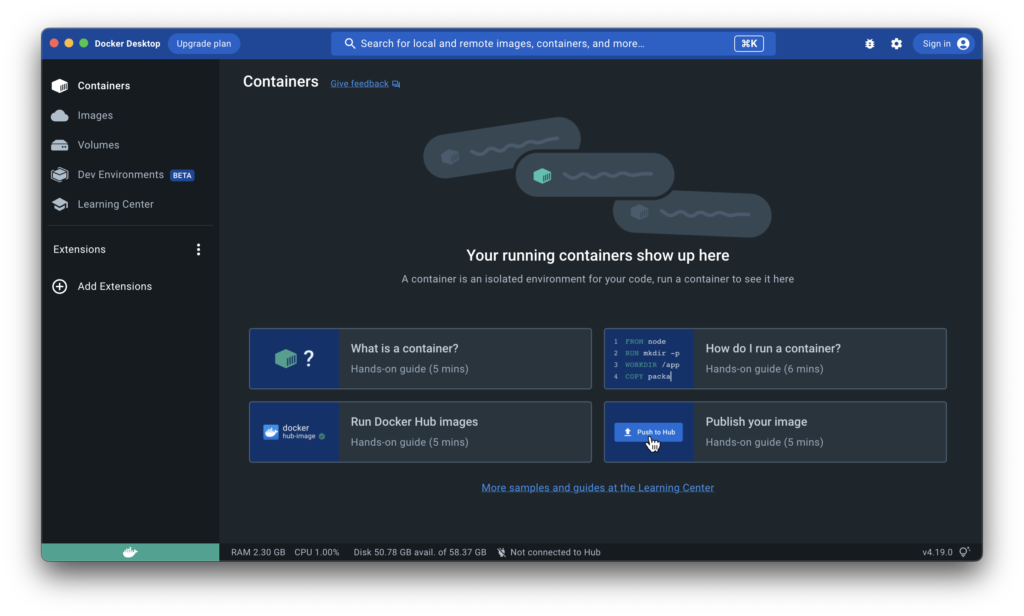
バイオインフォマティクスの分野ではCPUやメモリを多く使う処理も多いため、「設定(歯車アイコン)」→「Resources」の画面からDockerに割り当てられるCPUやメモリを確認しておきましょう。
Rosetta2のインストール
Rosetta2をインストールします。この部分は細かい話になるので詳細を理解する必要はないですが、設定しておくことをおすすめします。M1 Macは少し特殊なので設定をおこなっていると思ってください。
ターミナルで以下のコマンドを実行することで、Rosetta2をインストールできます。
softwareupdate --install-rosettaRosetta2のインストールが終わったら、Docker Desktop上で設定をおこないます。
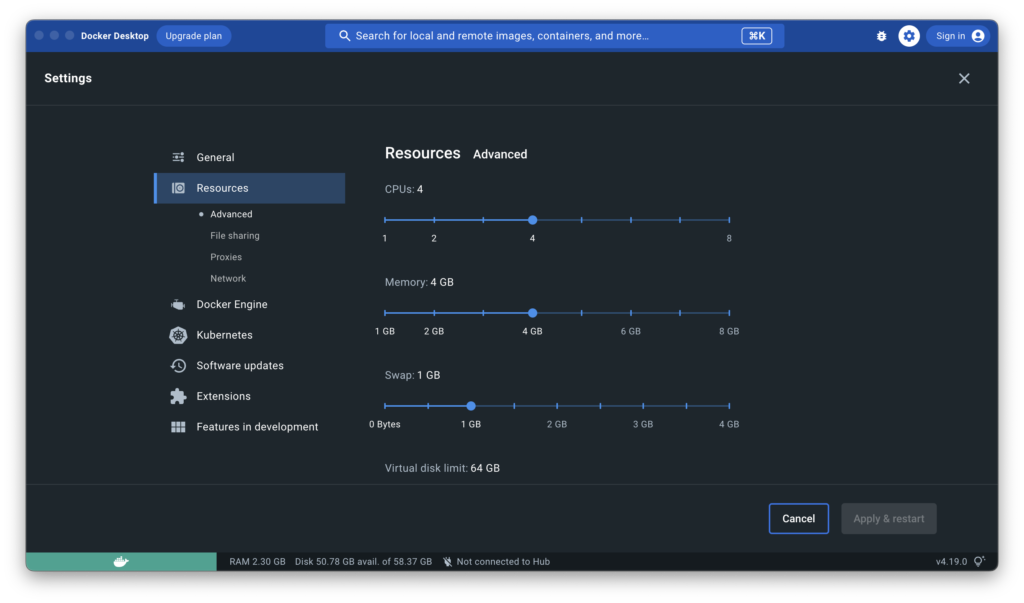
まず、「設定(歯車アイコン)」→「General」にある、「Use Virtualization framework」にチェックを入れます。
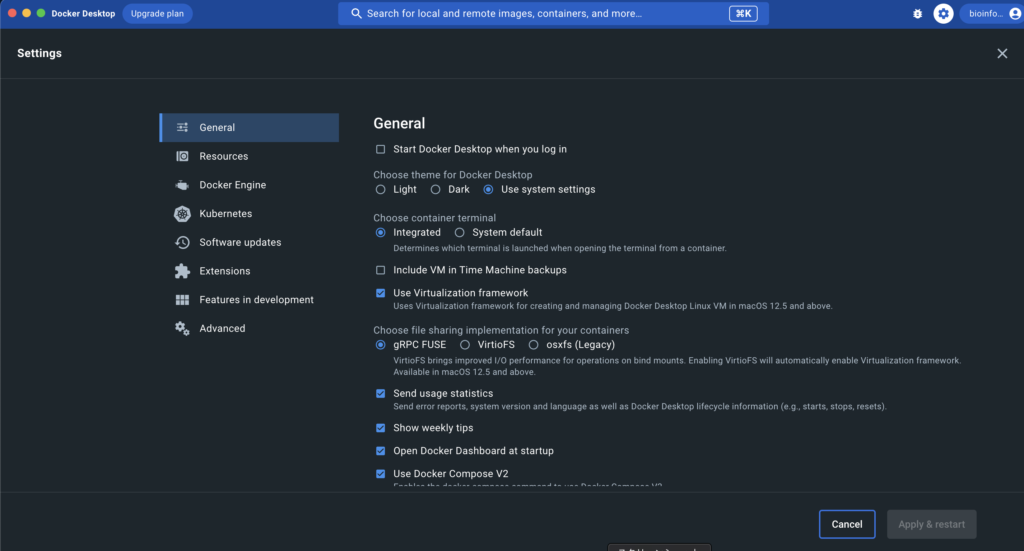
次に、「設定(歯車アイコン)」→「Features in development」にある、「Use Rosetta for x86/amd64 emulation on Apple Silicon」にチェックを入れ、「Apply & restart」をクリックします。
これで設定は完了です。
Dockerの使い方
これでDockerが使える状態となりました!
早速、Dockerを使用してコンテナを作成・起動してみましょう。
Python環境が整ったコンテナを使用するには、ターミナルから以下のようにコマンドを実行します。
docker container run -it --rm pythonすると、以下のようにpythonの対話モードが開始されます。ここで、-itは標準入出力をコンテナに結びつける(キー入力する場合)、–rmは使い終わったらコンテナを削除するオプションです。コンテナは使い終わったら削除するのが基本です。
Python 3.11.3 (main, May 23 2023, 08:45:42) [GCC 10.2.1 20210110] on linux
Type "help", "copyright", "credits" or "license" for more information.
>>> これだけでPython環境を用意することができました!
使い終わったら以下のコマンドでpythonの対話モードを終了させましょう。
>>> exit()DockerイメージからSRA Toolkitを使う
実際にバイオインフォマティクスのツールをDockerで使用してみましょう。
SRA Tookit
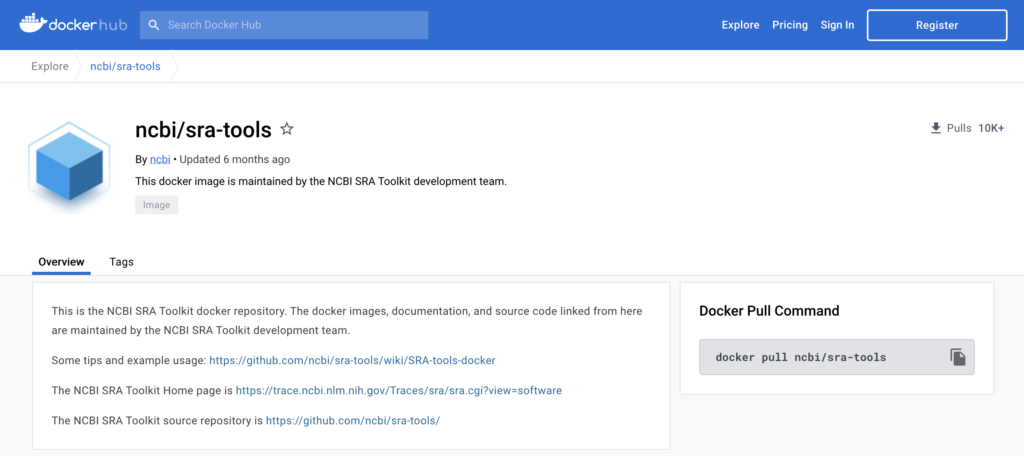
SRA Toolkitは公共データバンクのデータを扱うためのツールとライブラリパッケージです。dockerコンテナの設計図となる「イメージ」が公開されていますので、公開イメージを使用します。
以下のようなDocker Hubに登録されているイメージを使用します。
sra-toolsに含まれるfasterq-dumpコマンドでSRA(Sequence Read Archive)からシーケンスファイルをダウンロードし、FASTQ形式でデータを抽出します。
ターミナルから以下のように実行します。
docker container run --rm -v $PWD:/output -w /output ncbi/sra-tools:3.0.1 fasterq-dump -e 2 -p SRR000001うまくいけば、現在のフォルダにSRR000001.fastq, SRR000001_1.fastq, SRR0000001_2.fastqができているはずです。
Dockerを使わない場合、バイナリをダウンロードしてきて、パスを通すなど色々と面倒ですが、dockerを使うことで簡単にツールを利用することができました。
最後に
Dockerを使うことで、簡単に環境を作成したり、削除したりできます。
ぜひ活用することで様々なツールを試してみてください!